由福建农林大学牵头,联合武汉未来组及国内外二十多家科研机构在国际上率先完成了四倍体花生栽培种的全基因组测序工作,研究成果“The genome of cultivated peanut provides insight into legume karyotypes, polyploid evolution and crop domestication ”近日发表于国际著名专业期刊Nature Genetics。福建农林大学庄伟建教授、陈华博士、武汉未来组杨猛博士,以及美国佛罗里达大学博士生导师,福建农林大学兼职教授王建平博士为并列第一作者,其中武汉未来组李净净,梁帆,胡江,全伟鹏,樊俊鹏等为共同作者。本研究以狮头企(Arachis hypogaea var. Shitouqi)花生为材料,采用三代PacBio SMRT测序为主,结合Hi-C技术和高密度遗传图谱等完成了异源四倍体花生栽培种A、B亚基因组共20条染色体的精确组装,获得高质量的参考基因组。同时,对来自12个种的52份花生进行重测序,研究结果为花生的基因组结构、生物学特征、多倍体进化及作物驯化提供了新的见解。
花生是我国重要的油料作物,富含有益于心脑血管的油酸、亚麻油酸;白藜芦醇,纤维,叶酸和蛋白质等营养物质,被称作“长寿果”。在我国,花生的产量大约3,649千克每公顷,其贡献的产油量占所有油料作物的46%以上,经济价值位于水稻、小麦和玉米后,位于第四位。花生属包含81个种,大多为二倍体(2n = 2x = 20),而栽培种花生(Arachis hypogaea L.)为异源四倍体(AABB,2n = 4x = 40)。细胞遗传学,系统地理学和分子学证据表明,异源四倍体A. hypogaea可能是二倍体A. duranensis(AA)和A. ipaensis(BB)杂交形成,其基因组是野生二倍体的两倍。亚基因组之间的密切关系和高比例的重复序列增加了栽培花生基因组的组装难度。
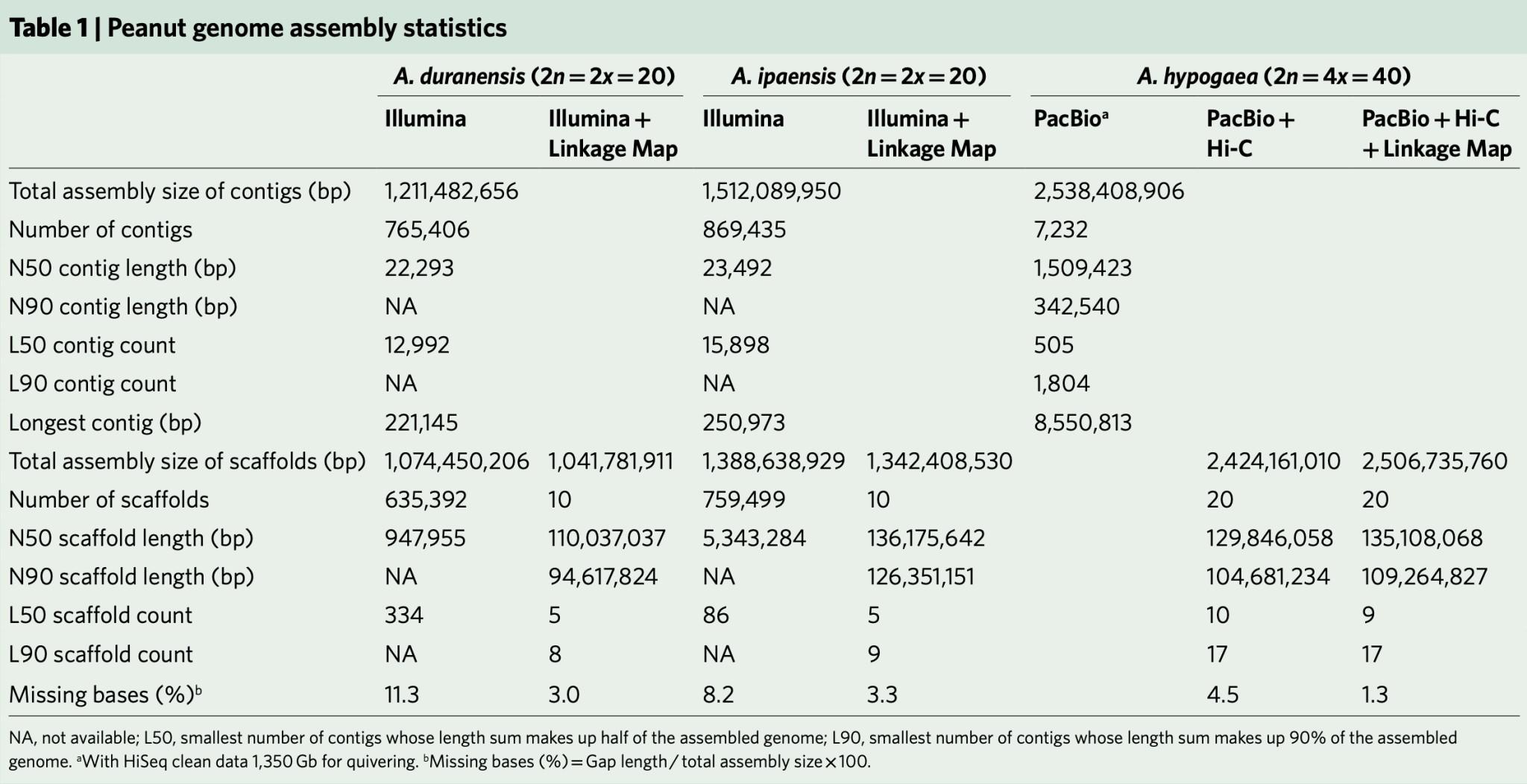
对100x PacBio数据进行初步组装(平均读长10.25Kb),获得Contig N50为1.51Mb,基因组大小2.54Gb,为预估基因组的94%。接着利用Hi-C数据进行聚类、纠错、排序,将PacBio Contig挂载到20个scaffolds,N50为129.8 Mb,使组装结果达到了染色体水平,包含95.5%的装配序列。最后利用ALLMAPS将四个高密度遗传图谱整合为包含14,619个标记、覆盖3,264 cM的遗传图谱,并基于此对5个含有轻微组装错误的Hi-C结果进行调整,最终组装出四倍体栽培种花生的20条染色体(Chr01-Chr20,对应野生祖先A基因组的A01-A10,以及B基因组的B01-B10),总大小为2.51Gb,占总组装长度的98.75%。为了评价组装效果,与公布的花生BAC双末端测序数据、三个花生全长BAC序列比对都显示高度的一致性,另外通过二代测序数据和三代的测序数据进行了碱基水平的准确性评估和连续性评估,所有的评估结果表明了花生基因组高质量组装。
表1 花生基因组组装统计
利用29个不同组织/条件的Illumina RNA-seq和PacBio Iso-Seq数据辅助注释,在组装的Shitouqi基因组中共预测到83,709个编码蛋白基因,其中功能注释基因占76.6%。在1,440个来自BUSCO数据库的基因集中,有93.1%在组装的结果中鉴定到,表明花生基因组高质量的组装和注释结果。
从花生基因组中共鉴定到30,596个非冗余基因,24,208个同源基因对在两个亚基因组之间表现出广泛的差异表达,其中B亚组的显性表达频率高于A亚组。
比较基因组结果表明花生栽培种B亚组与二倍体A. ipaensis一致性高于A亚组与A. Duranensis之间的一致性。共有629个基因受到基因转换的影响,有58.7% B转换为A,41.3% A转换为B。A和B亚组之间存在较多的倒转和重组,鉴定到至少6个有明确界限的A、B亚基因组之间的交换或替换,包括染色体3和13之间的10Mb易位。
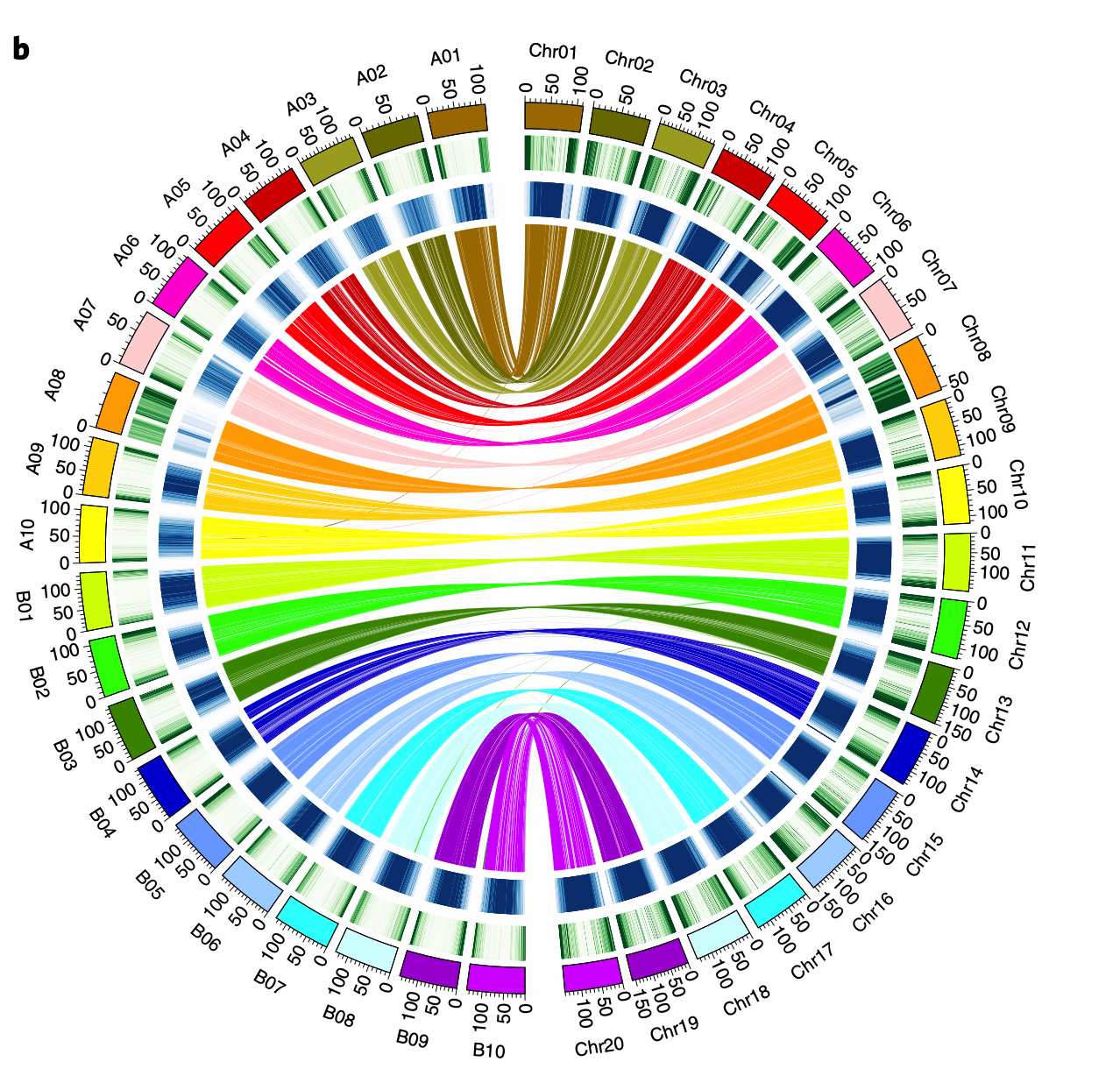
图1 花生亚基因组与二倍体A、B基因组基因密度、重复序列共线性关系
基因组重复序列(1.97 Gb)占组装总大小的77.65%,其中反转录转座子Gypsy LTR和non-autonomousLTR分别占40.59%和27.14%。重复序列分析发现大多数转座因子,特别是Gypsy LTR和non-autonomous LTR在四倍体化后发生扩增。通过完整的LTR反转座子两端的LTR序列进行比对计算碱基替代率表明A亚基因组在四倍体化后(约25万年前)经历了快速的LTR扩增,而B亚基因组和两个二倍体的LTR在四倍体化前扩增,这可能是由于功能障碍表达的普遍存在或四倍体花生中亚基因组同源染色体的缺失造成的,作者在这里提出疑问:测序的二倍体野生花生A. duranensis是否就是A亚基因组的祖先?
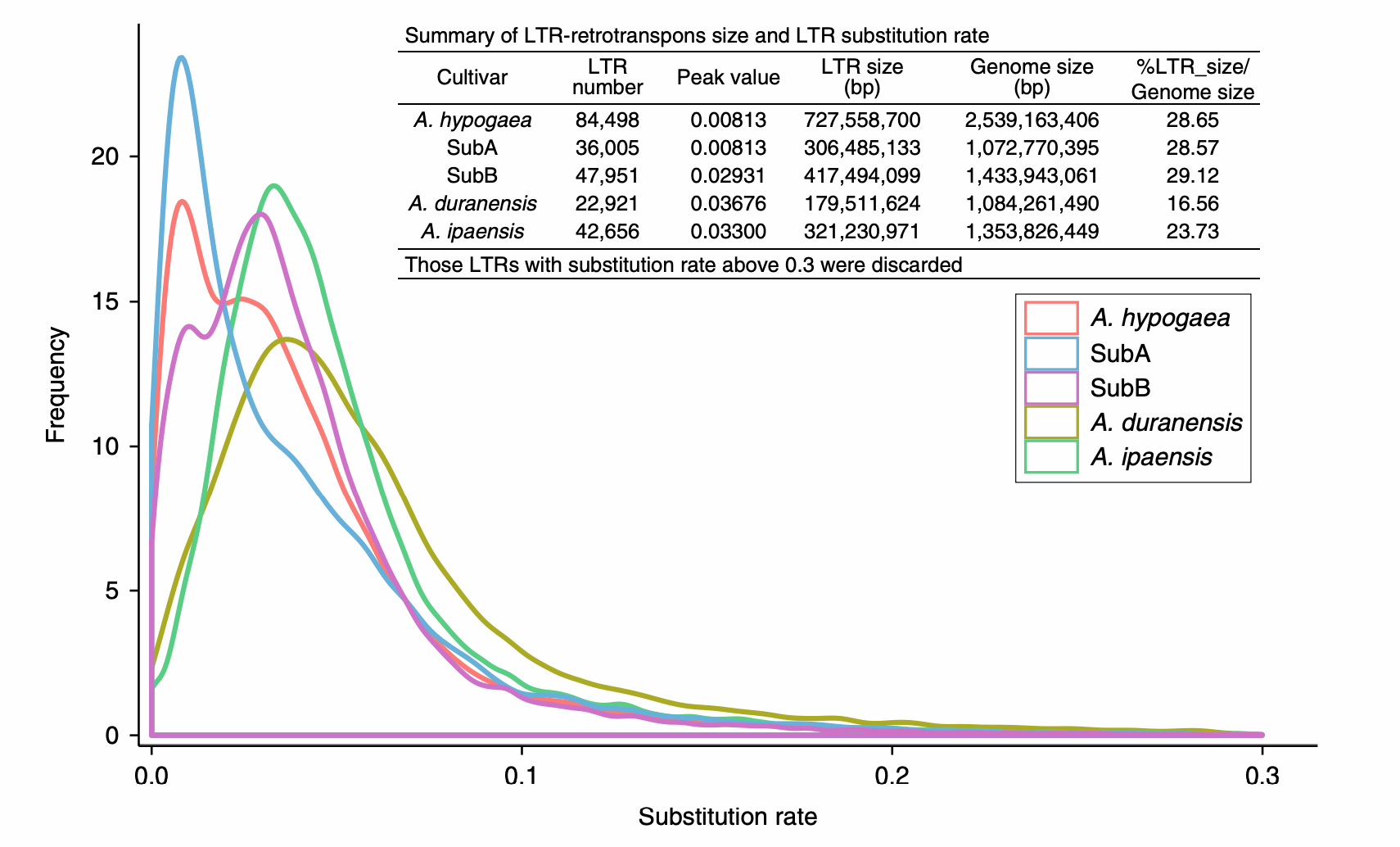
图2 花生及其二倍体祖先的重复序列扩增
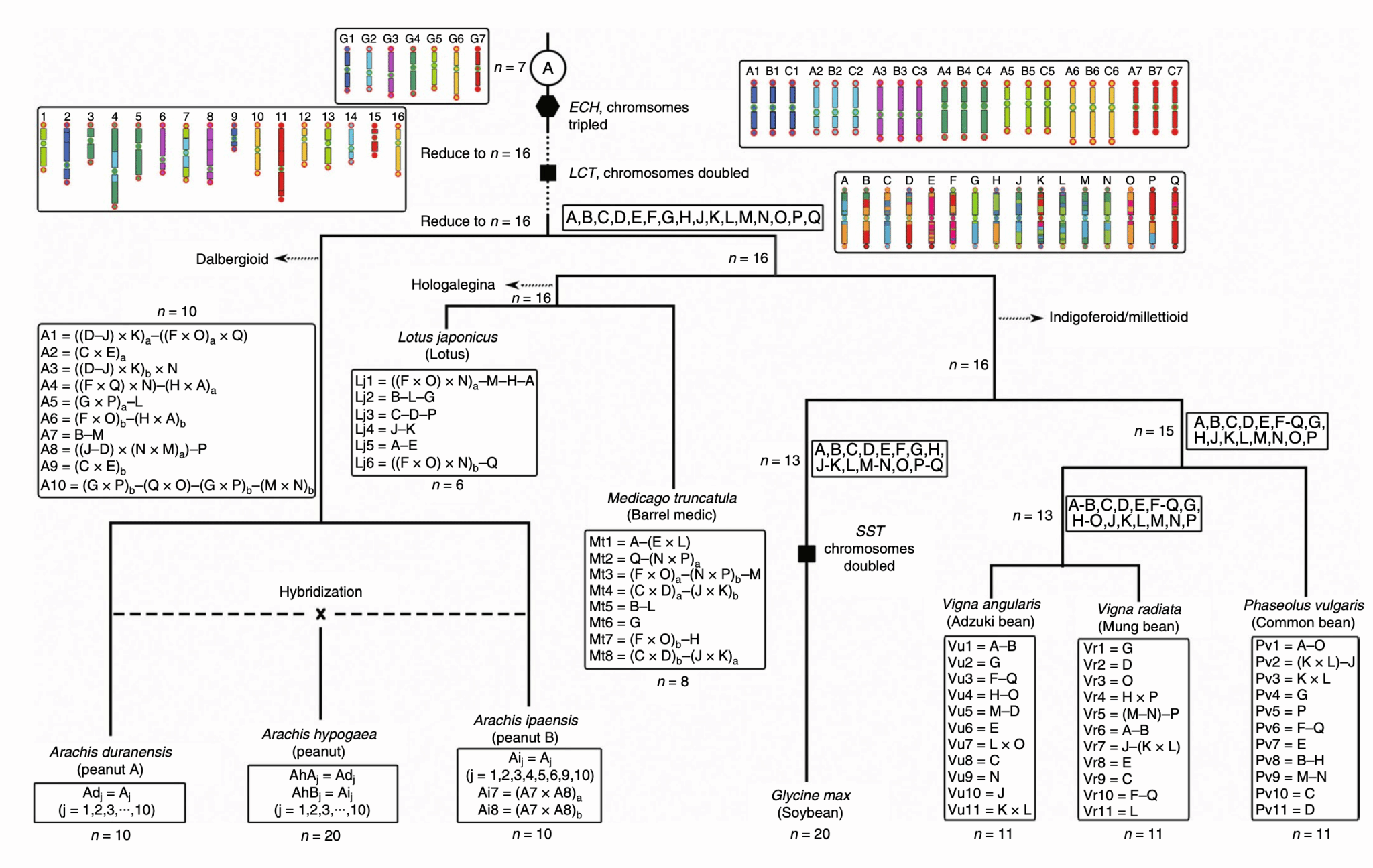
豆类植物共有的四倍化(legume-common tetraploidy,LCT;约5900万年前),以及主要双子叶植物共有的六倍化(core-eudicot-common hexaploidy,ECH;约1.3亿年前)痕迹保留在花生基因组中。作者利用保留有Post-ECH和post-LCT的普通豆类基因重建了16条原始豆类染色体(称为Lu),与现存的豆类基因组进行比较并绘制了花生与其他豆类的核型进化图,推断花生染色体的形成过程。花生祖先染色体A1,A3,A4,A5,A6和A7由Lu染色体经过6次融合造成染色体数目减少的片段组成;而A2,A8,A9和A10由两条Lu染色体的交叉互换产生;从A基因组分离以后,B基因组内的交叉互换形成了其特有的7、8号染色体。
与二倍体花生A. duranensis和A. ipaensis相比,四倍体花生亚基因组A(37,059 genes)和B(46,650 genes)分别有0.88%和12.46%的扩张,在A和B基因组二倍体中鉴定的24,380个同源基因家族中,90.68%在四倍化后仍旧保留。四倍体花生、野生A基因组和野生B基因组中的生长素响应因子(ARF)分别有114、28和28个,聚类为9个簇,其中Ⅰ-V仅包括四倍体花生的拷贝,同时花生含有3个CYP78A6(与种子生长有关),而二倍体B基因组中仅有一个拷贝,这可能与花生籽粒大小有关。
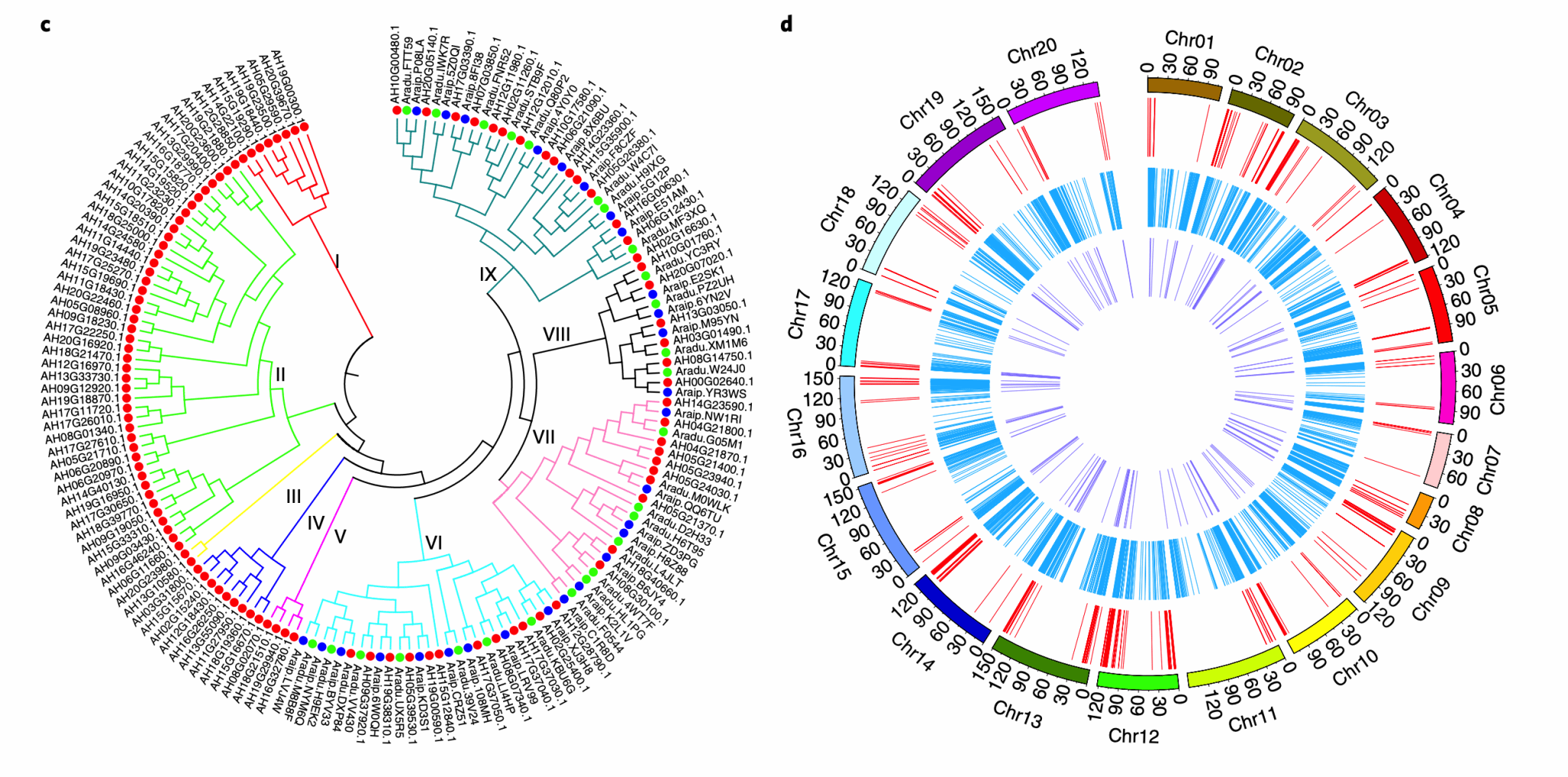
图4 生长素响应转录因子(ARF)家族进化树及脂肪酸代谢、氮共生途径及抗病基因染色体分布
驯化过程中同样会出现基因丢失的现象,例如四倍体花生有661个NBS结构的抗病基因,总数少于A. duranensis(385)和A.ipaensis(428)的总和,造成四倍体花生抗病基因的减少。作者还构建了花生基因组水平的酰基-脂质代谢网络和共生(SYM)信号通路基因的系统发育树,为花生品质改良及固氮研究提供支持。
花生起源于南美洲,被认为是A和B基因组A.duranensis和A.ipaensis之间的杂交,与二倍体A、B基因组比较,四倍体花生B亚基因组与A.ipaensis之间同源性在99.5%以上,而A亚基因组与A.duranensis之间仅有约97%的同源性。Ks 分布表明A、B基因组的分化预计在260万年前,与前人报道相同,而二倍体分化产生四倍体A或B基因组约在42-47万年前,要比之前认为的更古老(图5a)。
为了研究花生的起源和驯化,作者构建了52份样品(30个不同生态型异源四倍体花生,18个野生种,4个合成四倍体)的系统发育树(图5b)。系统发育树及测序数据表明野生型四倍体A.monticola形成了subsp. hypogaea和fastigiata生态型,这表明花生可能起源于不同的subsp. hypogaea并且在不同地点独立驯化,例如秘鲁西北地区进化出适应干旱的生态型(图5d,箭头B),东南独立驯化产生的瓦伦西亚和西班牙生态型在世界范围传播(图5d,箭头C和D)。这有别于前人预测的花生由A. monticola在阿根廷北部驯化而来。
四个合成四倍体中ISATGR 278和ISATGR 5发生了全基因组的加倍,而另外两个的A基因组分别是B基因组的1.23和5.93倍,这可能是由于亲本染色体由于不相容而在后代中不随机保留,这进一步支持了作者的假设:存在另一个与B基因组更相容的A基因组供体,而不是A. duranensis。
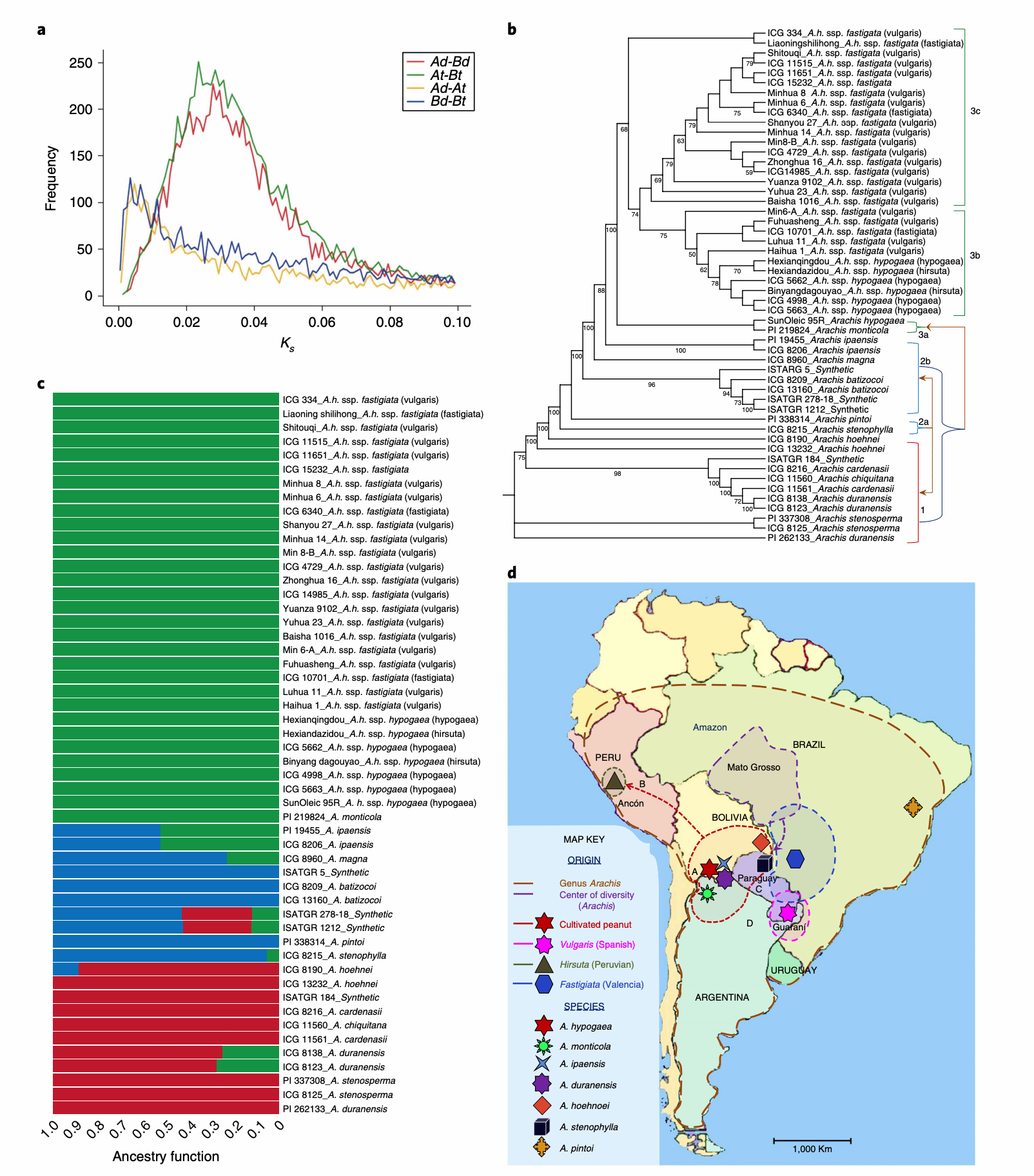
图5 花生的进化历史
该基因组揭示了许多已被基因定位的花生重要农艺性状的候选基因。控制红色种皮的单显性基因定位到3号染色体上一段0.905cM的区间内,包含WRKYs, MYB和bHLH家族以及细胞色素P450等花青素合成相关基因,这些基因的上调表达可能是红色种皮形成的原因。花生种子大小是重要的产量指标,作者利用一个重组自交系群体结合BSA分析在chr07和chr12染色体上定位到两个相同的候选区段,分别包含99和97个候选基因。基于高质量基因组的候选基因功能分析可以为花生种子大小调控提供许多新的信息。花生叶锈病和晚叶斑病(late leaf spot, LLS)共定位在同一基因组区域,重组自交群体抗病和感病池在Chr13染色体上显示重叠区域,进一步分析表明该区段内保守的Tir-NBS-LRR基因AH13G54010.1可能是两种病害的抗病基因。而有研究者利用二倍体A. duranensis基因组定位的叶锈病和晚叶斑病抗性区段位于Aradu.A03染色体,作者推测该区域可能是四倍化后从Chr03转移至Chr13。从含油量约40%的材料中获得了含油量高达80%的突变系,并通过重测序结合四倍体组装基因组解释了高含油量是由于ahFAD2A和ahFAD2B两种突变共同引起的。
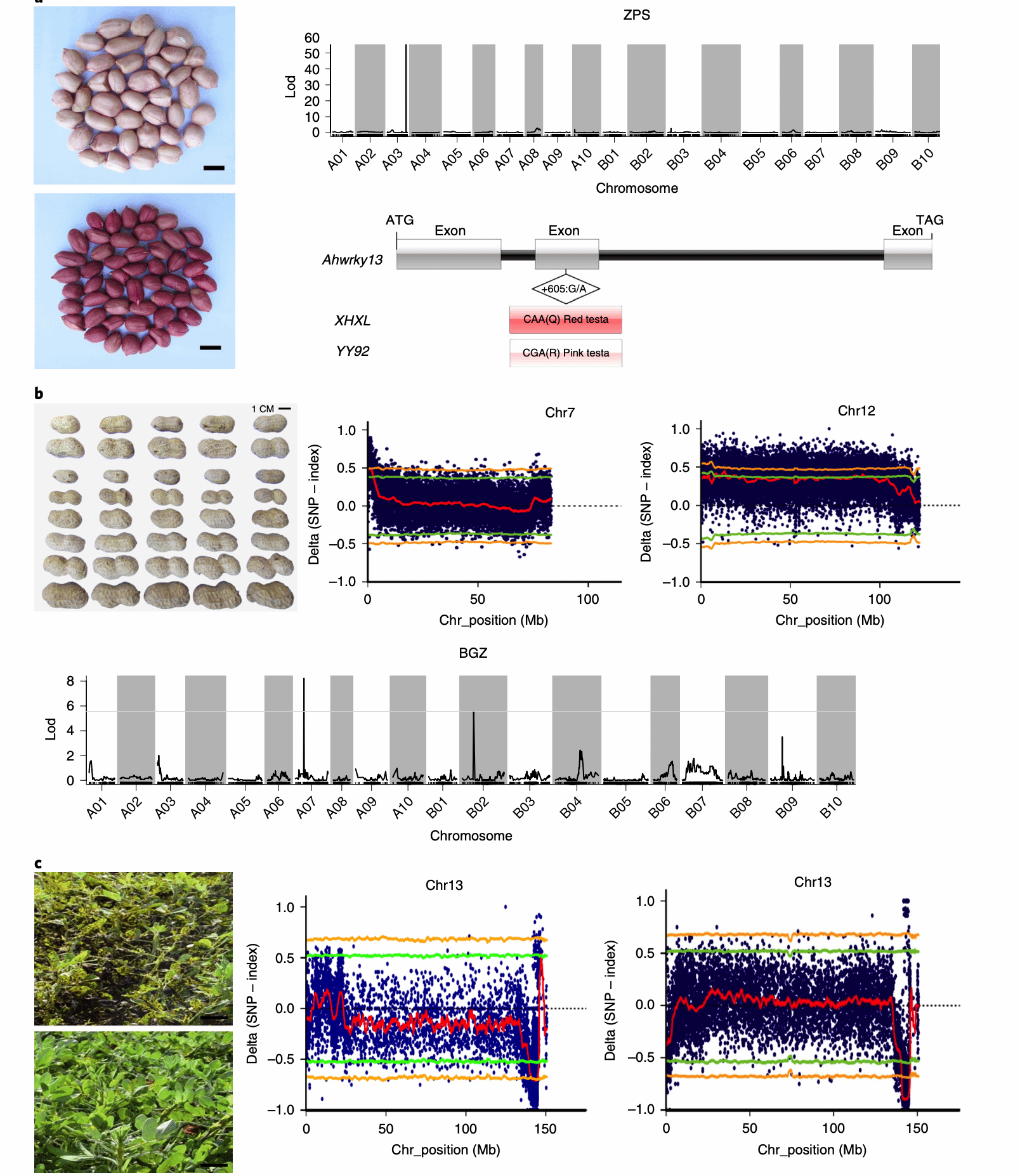
图6 种子大小、颜色和叶片抗病性的候选基因
本研究以中国花生品种狮头企为材料,组装出了染色体水平的高质量花生基因组,组装基因组大小2.54 Gb,包含20条染色体和83,709个蛋白编码基因。利用该高质量基因组,对种子大小进化、种子含油量、抗病性和共生固氮等功能基因家族进行了研究。
比较基因组分析表明相比A亚基因组,花生B亚基因组具有更多的基因和普遍的表达优势,这可能与A亚基因组中LTR的扩增有关,这也引出了A基因组起源的问题,即存在另一个与B基因组更相容的A基因组供体,而不是A. duranensis。
在A. hypogaea和其他豆类染色体进化方面,利用普通豆类基因重建了16条原始豆类染色体(称为Lu),与现存的豆类基因组进行了比较绘制了花生与其他豆类的核型进化图,为花生染色体的形成过程提供新的思路。
在花生起源于进化方面,52份种质材料的重测序分析表明花生可能起源于不同的subsp. hypogaea并且在不同地点独立驯化。
该高质量基因组揭示了许多花生重要农艺性状的候选基因,如种子大小、颜色、叶片抗病性等,将为为后续的功能基因组学研究和花生性状改良提供有意义的线索和数据支持。
参考文献:
Zhuang WJ, Chen H, Yang M, et al., The genome of cultivated peanut provides insight into legume karyotypes, polyploid evolution and crop domestication. Nature Genetics. 2019.
https://doi.org/10.1038/s41588-019-0402-2